การตรวจพิสูจน์บุคคลโดยใช้ SNP marker และการประยุกต์ในสิ่งส่งตรวจที่มีการเสื่อมสภาพของ DNA
พ.ต.ท.หญิง หทัยชนก บุญญฤทธิ์ นักวิทยาศาสตร์ (สบ 3) กลุ่มงานตรวจเลือดชีวเคมีและเขม่าดินปืน สถาบันนิติเวชวิทยา โรงพยาบาลตำรวจ
ปัจจุบันนี้การตรวจวิเคราะห์ DNA เพื่อพิสูจน์เอกลักษณ์บุคคลทางนิติเวชในประเทศไทยมีบทบาทอย่างมากในกระบวนการยุติธรรมไม่ว่าจะเป็นการพิสูจน์บุคคลมีชีวิต เศษชิ้นส่วนศพ โครงกระดูก ตลอดจนคราบต่าง ๆ ที่เกิดจากเนื้อเยื่อหรือสารคัดหลั่งจากมนุษย์ซึ่งก่อให้เกิดประโยชน์อย่างมากในการระบุตัวบุคคล ทั้งนี้ในการตรวจพิสูจน์บุคคลด้วยเทคนิค DNA นั้น มีการศึกษาค้นคว้าเพื่อหาตำแหน่งของ DNA ที่เหมาะสมที่จะนำมาใช้ในการตรวจพิสูจน์ ซึ่งในปัจจุบันการวิเคราะห์ STRs (Short Tandem Repeats) หรือ microsatellitesถือได้ว่าเป็น DNA marker มาตรฐานที่ใช้กันอย่างแพร่หลายทั้งภายในประเทศและต่างประเทศ อย่างไรก็ตาม ในกรณีภัยพิบัติ เช่นเหตุการณ์สึนามิถล่ม หรือกรณีคดีอาชญากรรมที่เกิดขึ้นแล้วหลายวันกว่าจะมีผู้พบศพ หรืออาจจะผ่านไปเป็นเดือน หรือปี หรือกรณีพบศพในสภาพแวดล้อมที่อำนวยต่อการเสื่อมสลายของ DNA เช่น ศพจมน้ำเสียชีวิต เป็นต้น ซึ่งสิ่งส่งตรวจจากที่เกิดเหตุเหล่านี้ไม่สามารถตรวจพิสูจน์ DNA โดย STR marker ได้เนื่องจาก DNA มีการเสื่อมสภาพหรือแตกเป็นชิ้นส่วนสั้น ๆ ที่มีขนาดน้อยกว่า 200 คู่เบส จึงไม่สามารถระบุเอกลักษณ์บุคคลนั้น ๆ และไม่สามารถสรุปผลได้
การตรวจพิสูจน์เอกลักษณ์บุคคลในกรณีสิ่งส่งตรวจที่มีการเสื่อมสภาพของ DNA จึงมีการศึกษาค้นคว้า SNP marker เพื่อใช้เป็นอีกทางเลือกหนึ่งเมื่อการใช้ STR marker อย่างเดียวไม่สามารถทำได้สนิป หรือ Single Nucleotide Polymorphisms (SNPs) เป็นความหลากหลายทางพันธุกรรมที่มีอยู่มากมายในจีโนมมนุษย์ (ซึ่งมีประมาณ 3 พันล้านเบส) ซึ่งเป็นความแตกต่างของลำดับเบสบนสายนิวคลีโอไทด์เพียงตำแหน่งเดียว เช่น การเปลี่ยนลำดับเบสบน DNA จาก AAGCT เป็น ATGCTอย่างไรก็ตาม ความแตกต่างที่เกิดขึ้นในจีโนมมนุษย์จะจัดเป็นสนิปก็ต่อเมื่อพบในประชากรมากกว่า 1% แต่หากน้อยกว่า 1% จะถือว่าเป็นการกลายพันธุ์เฉพาะจุด (point mutation) เท่านั้น ข้อได้เปรียบของ SNP marker เพื่อใช้ในการตรวจพิสูจน์เอกลักษณ์บุคคลทางนิติเวชคือ สามารถใช้ได้ดีกับสิ่งส่งตรวจที่มีการเสื่อมสภาพของ DNA เนื่องจาก SNP marker เป็นความหลากหลายที่ลำดับเบส 1 ตำแหน่ง ดังนั้น การออกแบบ primer เฉพาะเจาะจงที่ใช้ในการเพิ่มปริมาณ DNA ที่บริเวณ SNPsนั้น ๆ จะได้ PCR products ที่สั้นกว่า PCR productsที่เกิดจากการเพิ่มปริมาณ DNA ที่บริเวณ STR กล่าวคือ PCR productsจาก SNPsจะมีขนาด 60-120 คู่เบส โดย SNP marker ที่คัดเลือกมานั้นจะต้องให้ค่า Power of Discrimination (หรือ PD ซึ่งเป็นค่าที่บอกถึงกำลังในการแยกแยะบุคคล) เทียบเท่ากับ STR marker รวมทั้งต้องให้ค่า PD มากกว่า STR marker ในกรณีที่มีการเสื่อมสภาพของ DNA
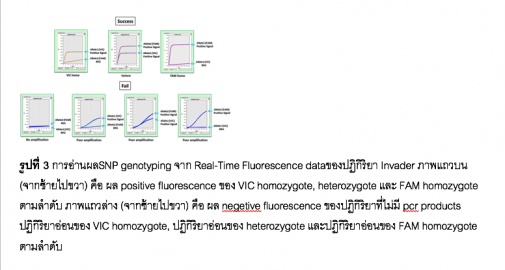
จากรายงานการวิจัยของ H. Boonyarit, et al. (2014) ได้ศึกษาวิจัยและจัดตั้งระบบ SNP marker ที่ใช้ในการตรวจพิสูจน์เอกลักษณ์บุคคลในกรณีสิ่งส่งตรวจที่มีการเสื่อมสภาพของ DNA ซึ่งสามารถใช้สนับสนุนผลการตรวจพิสูจน์เอกลักษณ์บุคคลด้วย STR marker งานวิจัยนี้ได้นำเสนอและรายงานผลวิธีที่นำมาใช้ในการตรวจวิเคราะห์ DNA ที่มีการเสื่อมสภาพโดยการตรวจดู SNP marker โดยคณะผู้วิจัยได้ค้นพบ SNP marker ที่เหมาะสมในการแยกแยะบุคคลในประชากรไทยที่กระจายอยู่บนทุกโครโมโซมจำนวน 54 ตำแหน่ง (รูปที่ 1) และได้ทำการพัฒนาวิธีการตรวจ SNP marker เหล่านั้นให้สะดวกต่อการนำไปใช้งานจริงในห้องปฏิบัติการที่มีเครื่องมือที่ไม่ซับซ้อน ซึ่งในการพัฒนาวิธีการตรวจ SNP marker เพื่อให้สามารถใช้ได้ดีกับสิ่งส่งตรวจที่มีการเสื่อมสภาพของ DNA หรืออย่างน้อยสามารถใช้ SNP marker สนับสนุน STR marker ได้ในกรณีการตรวจพิสูจน์เอกลักษณ์บุคคลทางนิติเวชที่มีการเสื่อมสภาพของ DNA ในงานวิจัยนี้ได้มีการออกแบบให้ได้ PCR product มีขนาดสั้นกว่า 100 เบส โดยที่ค่าเฉลี่ยของขนาด PCR product ในงานวิจัยนี้คือ 71 เบส (ขนาดตั้งแต่ 51-98 เบส) และใช้วิธีตรวจสอบPCR product ด้วยวิธี Invader assay (รูปที่ 2) ดังนั้น primary probes และ Invader oligonucleotides ที่ใช้ในการตรวจสอบ SNP marker แต่ละตำแหน่งจะถูกออกแบบและสังเคราะห์ตามหลักการของวิธี Invader assay จากนั้นได้ทำการพัฒนาระบบ Multiplex PCR-based Invader assay ซึ่งปฏิกิริยาในการพัฒนา Multiplex PCR-based Invader assay ได้ถูกนำมาทดลองทำหลายครั้งจนในที่สุดได้ค่าที่ทำให้ได้ปฏิกิริยาที่ดีที่สุด และสุดท้ายได้แบ่งปฏิกิริยา PCR ออกเป็น 2 ชุด สำหรับการตรวจสอบ SNP markerจำนวน 54 ตัวนี้ โดยแบ่งเป็นชุดละ 30 ตัว และ 24 ตัว ตามลำดับ จากนั้นนำ PCR products ที่ได้จากปฏิกิริยา PCR 2 ชุดนี้ไปทำปฏิกิริยา Invader ต่อไป โดยใช้ multiplex PCR products เป็นตัวตั้งต้นในปฏิกิริยา Invader หลังจากนั้นทำการวิเคราะห์ผลโดยการสร้าง real-time signal intensity plot ด้วยโปรแกรม Sequence Detection System (SDS) 2.2 software (Applied Biosystems, Foster City, CA, USA) (รูปที่ 3)
ขั้นตอนสุดท้ายคือ การดำเนินการเปรียบเทียบผลการวิเคราะห์ทางสถิติที่ได้จากวิธีการตรวจ SNP genotyping กับวิธีการตรวจมาตรฐาน STR genotyping ซึ่งจากผลการวิเคราะห์เปรียบเทียบพบว่า การตรวจ พิสูจน์เอกลักษณ์บุคคลโดยใช้ 54-SNP marker set ที่พัฒนาได้นี้มีความสามารถในการแยกแยะบุคคลใน ประชากรไทยสูงกว่าวิธีมาตรฐานที่ใช้อยู่ในปัจจุบัน และสามารถใช้ได้เป็นอย่างดีกับการตรวจพิสูจน์เอกลักษณ์ บุคคลในกรณีสิ่งส่งตรวจที่มีการเสื่อมสภาพของ DNA ที่การใช้ STR marker ไม่สามารถตรวจพิสูจน์เอกลักษณ์บุคคลได้ จะเห็นได้ว่าการจัดตั้งระบบ SNP marker ที่ใช้ในการตรวจพิสูจน์เอกลักษณ์บุคคลในกรณีสิ่งส่งตรวจมีการเสื่อมสภาพของ DNA นี้ สามารถใช้สนับสนุนผลการตรวจพิสูจน์เอกลักษณ์บุคคลด้วย STR marker ทั้งนี้ SNP marker จะเป็นทางเลือกที่เข้ามาสนับสนุนการตรวจพิสูจน์เอกลักษณ์บุคคลของผู้เสียชีวิตนั้น ๆ ได้ โดยอาจใช้ SNP marker ในการตรวจพิสูจน์เอกลักษณ์บุคคลในตัวอย่างที่มี DNA เสื่อมสภาพและไม่สามารถตรวจหารูปแบบ DNA ของ STR marker ได้ หรืออาจใช้ร่วมกับ STR marker ในการตรวจพิสูจน์เอกลักษณ์บุคคลในตัวอย่างที่มี DNA เสื่อมสภาพ และสามารถตรวจหารูปแบบ DNA ของ STR marker ได้เพียงบางตำแหน่งเท่านั้น ซึ่งโดยส่วนรวมก็จะมีประโยชน์ต่อสังคมคือ ได้มีการจัดตั้งระบบ SNP marker อีกระบบเพื่อใช้ในกรณี DNA มีการเสื่อมสภาพ
รูปที่ 1 ตำแหน่งบนโครโมโซมของ SNPs ที่เหมาะสมในการแยกแยะบุคคลในประชากรไทยที่กระจายอยู่บนทุกโครโมโซม จำนวน 54 ตำแหน่ง
รูปที่ 2 หลักการ Invader Assay ประกอบด้วย Invader probe 1 ตัว และ allele-specific probe 2 ตัวที่ติดฉลากสี fluorescence ต่างกัน 2 สี ได้แก่ R คือ reporter dye และ Q คือ quencher dye ในการเกิดปฏิกิริยา Invader นั้น probe ทั้ง 3 ตัวจะจับกับ target DNA เกิดเป็นโครงสร้างหรือรูปร่างแบบ 3-Dimension ตรงบริเวณที่เป็น SNPs ซึ่งโครงสร้างดังกล่าวนี้จะถูกจดจำโดย clevage enzyme ขั้นตอนต่อมา enzyme จะตัดสายโครงสร้างนี้ก็ต่อเมื่อ allele-specific probe มีเบสที่ complementary กับ SNPs และปลดปล่อยสีที่ reporter dye ซึ่งจะเป็นตัวบ่งชี้เบสที่อยู่บน SNPsตำแหน่งนั้น ๆ
รูปที่ 3 การอ่านผลSNP genotyping จาก Real-Time Fluorescence dataของปฏิกิริยา Invader ภาพแถวบน (จากซ้ายไปขวา) คือ ผล positive fluorescence ของ VIC homozygote, heterozygote และ FAM homozygote ตามลำดับ ภาพแถวล่าง (จากซ้ายไปขวา) คือ ผล negetive fluorescence ของปฏิกิริยาที่ไม่มี pcr products ปฏิกิริยาอ่อนของ VIC homozygote, ปฏิกิริยาอ่อนของ heterozygote และปฏิกิริยาอ่อนของ FAM homozygote ตามลำดับ
เอกสารอ้างอิง
- H. Boonyarit, S. Mahasirimongkol, N. Chavalvechakul, M. Aoki, H. Amitani, N. Hosono, N. Kamatani, M. Kubo, P. Lertrit. Development of a SNP set for human identification: A set with high powers of discrimination which yields high genetic information from naturally degraded DNA samples in the Thai population. J Forensic Sci 2014;11:166-73.
- M. Kayser, PD. Knijff. Improving human forensics through advances in genetics, genomics, and molecular biology. Nat Genet Rev 2011;12:179-92.
- M. Fondevila, C. Phillips, N. Naveran, M. Cerezo, A. Rodriguez, R. Calvo, et al. Challenging DNA: assessment of a range of genotyping approaches for highly degraded forensic samples. Forensic Sci Int Genet Suppl Ser 2008;1:26-8.
- E. Giardina, I. Pietrangeli, C. Martone, P. Asili, I. Predazzi, P. Marsala, et al. In silico and in vitro comparative analysis to select, validate and test SNPs for human identification. BMC Genom 2007;457-63.
- E. Giardina, I. Predazzi, I. Pietrangeli, P. Asili, P. Marsala, L. Gabriele, et al. Frequency assessment of SNPs for forensic identification in different population. Forensic Sci Int Genet 2007;1:e1-e3.
- SNP genotyping: Technologies and Biomedical Applications. Annu Rev Biomed Eng 2007;9:289-320.
- JJ. Sanchez, C. Phillips, C. Borsting, K. Balogh, M. Bogus, M. Fondevila, et al. A multiplex assay with 52 single nucleotide polymorphisms for human identification. Electrophoresis 2006;27:1713-24.
- P. Gill. An assessment of the utility of single nucleotide polymorphisms (SNPs) for Forensic purposes. Int J Legal Med 2001;114:204-10.